



使用keras搭建神经网络
阅读:1234658 分享到Keras 是一个高层神经网络库,Keras 由纯 Python 编写而成并基 Tensorflow 或 Theano。Keras 为支持快速实验而生,能够把你的 idea 迅速转换为结果。
Tensorflow 2.0 已经与 Keras 深度进行了融合,我们既可以用 Keras 的高级接口迅速搭建和训练模型。
六步骤法
使用 keras 搭建神经网络遵循 6 步法:
第1步(import):import 需要的模块。
第2步(train,test):告知要喂入网络的训练集和测试集是什么,也就是训练集的特征值和标签(x_train,y_train)以及训练集的特征值和标签(x_test,y_test)。
第3步(model=tf.keras.models.Squential):在 Sequential 中搭建网络结构,逐层描述每层网络,也就是实现前向传播。
第4步(model.compile):在 compile 中配置训练方法,告知训练时使用哪种优化器,选择哪个损失函数,选择哪种评测指标。
第5步(model.fit):在fit中执行训练过程,告知训练集和测试集的输入特征和标签,告知每个 batch 是多少,告知要迭代多少次数据集。
第6步(model.summary):使用 summary 打印出网络的结构和参数统计。
keras 函数详解
以后我们搭建神经网络就按照上面的六步法则进行编写代码,接下来我们来学习 keras 提供的这些函数的用法。
Sequential 可以认为是个容器,这个容器里面封装了一个神经网络结构,我们可以使用 Sequential 描述从输入层到输出层每一层的网络结构,每一层的网络结构可以是一下几种。
1.拉直层:tf.keras.layers.Flatten()。有时候我们从第三方机构获取的数据集特征值是二维的(比如图片特征值),我们可以使用该函数把矩阵转换为一维向量。
2.全连接层:tf.keras.layers.Dense(神经元个数,activation=“激活函数”,kernel_regularizer=正则化)。其中 activation 以字符串给出,可以使用的激活函数有 relu、softmox、sigmod、tanh等;其中 kernel_regularizer 填入函数名,我们可以使用 l1 正则化和 l2 正则化,分别使用函数 tf.eras.regularizers.l1()、tf.keras.regularizers.l2()。
3.卷积层:tf.keras.layers.Conv2D(filers=卷积核个数,kernel_size=卷积核尺寸,strides=卷积步长,padding=“valid”or“same”)。卷积层的知识我们后面再介绍。
4.LSTM层:tf.keras.layers.LSTM()。循环网络层的知识我们后面再介绍。
Model.compile(optimizer=优化器,loss=损失函数,metrics=[“准确率”])函数配置神经网络的训练方法,我们在 compile 函数的参数中设置训练时选择的优化器、损失函数和评测指标,其中优化器和损失函数可以是以字符串的形式给出也可以以函数的形式给出。
优化器 Optimizer 的选择有以下几种:
1.‘sgd’ or tf.keras.optimizers.SGD (lr=学习率,momentum=动量参数)
2.‘adagrad’ or tf.keras.optimizers.Adagrad (lr=学习率)
3.‘adadelta’ or tf.keras.optimizers.Adadelta (lr=学习率)
4.‘adam’ or tf.keras.optimizers.Adam (lr=学习率, beta_1=0.9, beta_2=0.999)
损失函数 loss 的选择有以下几种:
1.‘mse’ or tf.keras.losses.MeanSquaredError()
2.‘sparse_categorical_crossentropy’ or tf.keras.losses.SparseCategoricalCrossentropy(from_logits=False)
使用 keras 高阶 API 训练神经网络时,我们常使用第二种损失函数,该函数有个 from_logits 参数,如果神经网络使用了 softmax 使得输出符合概率分布或使用 sigmod 使得输出分布在 [0,1] 之间,该参数设置为 False,否则该参数设置为 True。
网络评测指标 metrics 的选择有以下几种:
1.‘accuracy’:y_和y都是数值,如y_=[1] y=[1]
2.‘categorical_accuracy’:y_和y都是独热码(概率分布),如y_=[0,1,0] y=[0.256,0.695,0.048]
3.‘sparse_categorical_accuracy’:y_是数值,y是独热码(概率分布)或(0, 1)之间的值,如y_=[1] y=[0.256,0.695,0.048]
我们从第三方获取的数据集大都是以数值形式给出标签的值,比如前面使用的鸢尾花数据集的标签,而我们在训练神经网络时,对于单分类问题我们通常都会把前向传播的输出使用 softmax 激活函数映射为符合概率分布的结果,对于多分类问题我们一般则使用 sigmod 激活函数把前向传播的输出映射到(0, 1)之间,所以 metrics 参数的值我们一般都是用第 3 种;如果你想使用第 2 种,则需要自己使用 one_hot 函数把标签转为独热码形式。
model.fit(训练集的输入特征,训练集的标签,batch_size=,epochs=,validation_data=(测试集的输入特征,测试集的标签),validation_split=从训练集划分多少比例给测试集,validation_freq=多少次epoch测试一次),其中 validation_data 和 validation_split 只需要使用一个。
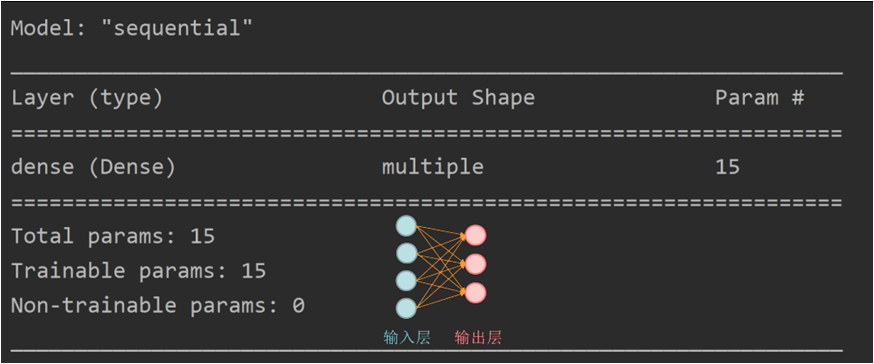
model.summary 可以打印出网络的结构和参数统计,如下图是我们前面的鸢尾花分类的输出结果,该网络结构为 4 个输入(4 个特征值),3 个输出(标签 3 分类)的一层网络(一个 w),dense 全连接共有 15 个参数(参数 w 是 4 行 3 列共 12 个元素,参数 b 是 3 个元素的向量,共 15 个参数),Trainable params 是可训练参数共有 15 个,Non-trainable 是不可训练参数有 0 个。
keras 实现鸢尾花分类
下面我们使用 keras 的 6 步法编写代码来实现鸢尾花分类。
import tensorflow as tf from sklearn import datasets import numpy as np x_train = datasets.load_iris().data y_train = datasets.load_iris().target np.random.seed(1024) np.random.shuffle(x_train) np.random.seed(1024) np.random.shuffle(y_train) ''' 1.目前我们使用1层神经网络,所以共有3个神经元(输出层3分类所以是3个神经元,输入层不做计算不是神经元) 2.激活函数使用softmax使得结果符合概率分布 3.正则化使用l2正则化 4.Sequential参数是个list,本案例我们只使用1层神经网络,所以list填入1个dense,如果你使用多层神经网络 则需要在list中增加隐藏层的dense。 ''' model = tf.keras.models.Sequential([ tf.keras.layers.Dense(3, activation="softmax", kernel_regularizer=tf.keras.regularizers.l2()) ]) ''' 1.优化器使用SGD,学习率设置为0.1 2.激活函数使用softmax使得结果符合概率分布,所以from_logits=False 3.鸢尾花数据集的标签是数值,我们前向传播结果是独热码符合概率分布 ''' model.compile(optimizer=tf.keras.optimizers.SGD(lr=0.1), loss=tf.keras.losses.SparseCategoricalCrossentropy(from_logits=False), metrics=["sparse_categorical_accuracy"]) ''' 1.x_train是训练特征值,y_train是训练标签 2.batch_size:每次喂入神经网络32组数据 3.epochs:迭代循环次数500 4.validation_split:从训练集的20%作为测试集 5.validation_freq:每迭代20次从测试集中验证一次准确率 ''' model.fit(x_train, y_train, batch_size=32, epochs=100, validation_split=0.2, validation_freq=20) # 打印出神经网络结构和参数 model.summary()
代码运行结果如下:
4/4 [==============================] - 0s 5ms/step - loss: 1.6351 - sparse_categorical_accuracy: 0.2417 Epoch 2/100 4/4 [==============================] - 0s 1ms/step - loss: 1.3186 - sparse_categorical_accuracy: 0.4250 Epoch 3/100 4/4 [==============================] - 0s 2ms/step - loss: 1.1365 - sparse_categorical_accuracy: 0.6583 Epoch 4/100 4/4 [==============================] - 0s 2ms/step - loss: 0.8764 - sparse_categorical_accuracy: 0.6667 Epoch 5/100 4/4 [==============================] - 0s 2ms/step - loss: 1.1204 - sparse_categorical_accuracy: 0.5500 Epoch 6/100 4/4 [==============================] - 0s 2ms/step - loss: 0.7333 - sparse_categorical_accuracy: 0.6417 Epoch 7/100 4/4 [==============================] - 0s 1ms/step - loss: 0.8290 - sparse_categorical_accuracy: 0.7000 Epoch 8/100 4/4 [==============================] - 0s 2ms/step - loss: 0.8973 - sparse_categorical_accuracy: 0.6333 Epoch 9/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6544 - sparse_categorical_accuracy: 0.6750 Epoch 10/100 4/4 [==============================] - 0s 2ms/step - loss: 0.6212 - sparse_categorical_accuracy: 0.7083 Epoch 11/100 4/4 [==============================] - 0s 1000us/step - loss: 0.7828 - sparse_categorical_accuracy: 0.6667 Epoch 12/100 4/4 [==============================] - 0s 2ms/step - loss: 0.9137 - sparse_categorical_accuracy: 0.7000 Epoch 13/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5994 - sparse_categorical_accuracy: 0.7167 Epoch 14/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4895 - sparse_categorical_accuracy: 0.9083 Epoch 15/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6755 - sparse_categorical_accuracy: 0.7167 Epoch 16/100 4/4 [==============================] - 0s 1ms/step - loss: 0.9959 - sparse_categorical_accuracy: 0.6500 Epoch 17/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6598 - sparse_categorical_accuracy: 0.8250 Epoch 18/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4618 - sparse_categorical_accuracy: 0.9250 Epoch 19/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4634 - sparse_categorical_accuracy: 0.8333 Epoch 20/100 4/4 [==============================] - 0s 108ms/step - loss: 0.6006 - sparse_categorical_accuracy: 0.6750 - val_loss: 0.4201 - val_sparse_categorical_accuracy: 0.8000 Epoch 21/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5555 - sparse_categorical_accuracy: 0.7083 Epoch 22/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4485 - sparse_categorical_accuracy: 0.8750 Epoch 23/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6362 - sparse_categorical_accuracy: 0.7000 Epoch 24/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5023 - sparse_categorical_accuracy: 0.8333 Epoch 25/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4327 - sparse_categorical_accuracy: 0.8667 Epoch 26/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4627 - sparse_categorical_accuracy: 0.8083 Epoch 27/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6103 - sparse_categorical_accuracy: 0.7000 Epoch 28/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5095 - sparse_categorical_accuracy: 0.8333 Epoch 29/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4225 - sparse_categorical_accuracy: 0.8917 Epoch 30/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4350 - sparse_categorical_accuracy: 0.8917 Epoch 31/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4355 - sparse_categorical_accuracy: 0.9167 Epoch 32/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5568 - sparse_categorical_accuracy: 0.7917 Epoch 33/100 4/4 [==============================] - 0s 750us/step - loss: 0.4678 - sparse_categorical_accuracy: 0.8667 Epoch 34/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6253 - sparse_categorical_accuracy: 0.7000 Epoch 35/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4059 - sparse_categorical_accuracy: 0.9583 Epoch 36/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5605 - sparse_categorical_accuracy: 0.7583 Epoch 37/100 4/4 [==============================] - 0s 2ms/step - loss: 0.5138 - sparse_categorical_accuracy: 0.7250 Epoch 38/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4304 - sparse_categorical_accuracy: 0.9000 Epoch 39/100 4/4 [==============================] - 0s 1ms/step - loss: 0.5555 - sparse_categorical_accuracy: 0.7417 Epoch 40/100 4/4 [==============================] - 0s 14ms/step - loss: 0.6257 - sparse_categorical_accuracy: 0.7750 - val_loss: 0.3507 - val_sparse_categorical_accuracy: 0.9667 Epoch 41/100 4/4 [==============================] - 0s 6ms/step - loss: 0.5206 - sparse_categorical_accuracy: 0.7500 Epoch 42/100 4/4 [==============================] - 0s 3ms/step - loss: 0.4249 - sparse_categorical_accuracy: 0.8250 Epoch 43/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4254 - sparse_categorical_accuracy: 0.8917 Epoch 44/100 4/4 [==============================] - 0s 5ms/step - loss: 0.8867 - sparse_categorical_accuracy: 0.5917 Epoch 45/100 4/4 [==============================] - 0s 2ms/step - loss: 0.7229 - sparse_categorical_accuracy: 0.6750 Epoch 46/100 4/4 [==============================] - 0s 4ms/step - loss: 0.5368 - sparse_categorical_accuracy: 0.7917 Epoch 47/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4386 - sparse_categorical_accuracy: 0.8583 Epoch 48/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4074 - sparse_categorical_accuracy: 0.9250 Epoch 49/100 4/4 [==============================] - 0s 4ms/step - loss: 0.3923 - sparse_categorical_accuracy: 0.9250 Epoch 50/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4920 - sparse_categorical_accuracy: 0.8167 Epoch 51/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4358 - sparse_categorical_accuracy: 0.8250 Epoch 52/100 4/4 [==============================] - 0s 2ms/step - loss: 0.5347 - sparse_categorical_accuracy: 0.7250 Epoch 53/100 4/4 [==============================] - 0s 3ms/step - loss: 0.3951 - sparse_categorical_accuracy: 0.9167 Epoch 54/100 4/4 [==============================] - 0s 3ms/step - loss: 0.4206 - sparse_categorical_accuracy: 0.8750 Epoch 55/100 4/4 [==============================] - 0s 12ms/step - loss: 0.4214 - sparse_categorical_accuracy: 0.8500 Epoch 56/100 4/4 [==============================] - 0s 14ms/step - loss: 0.3791 - sparse_categorical_accuracy: 0.9500 Epoch 57/100 4/4 [==============================] - 0s 5ms/step - loss: 0.4462 - sparse_categorical_accuracy: 0.8667 Epoch 58/100 4/4 [==============================] - 0s 16ms/step - loss: 0.3880 - sparse_categorical_accuracy: 0.9083 Epoch 59/100 4/4 [==============================] - 0s 19ms/step - loss: 0.4217 - sparse_categorical_accuracy: 0.8750 Epoch 60/100 4/4 [==============================] - 0s 105ms/step - loss: 0.3733 - sparse_categorical_accuracy: 0.9333 - val_loss: 0.4586 - val_sparse_categorical_accuracy: 0.7667 Epoch 61/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4252 - sparse_categorical_accuracy: 0.8667 Epoch 62/100 4/4 [==============================] - 0s 7ms/step - loss: 0.4210 - sparse_categorical_accuracy: 0.8583 Epoch 63/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4720 - sparse_categorical_accuracy: 0.8083 Epoch 64/100 4/4 [==============================] - 0s 1ms/step - loss: 0.3808 - sparse_categorical_accuracy: 0.9333 Epoch 65/100 4/4 [==============================] - 0s 2ms/step - loss: 0.5067 - sparse_categorical_accuracy: 0.7833 Epoch 66/100 4/4 [==============================] - 0s 3ms/step - loss: 0.4058 - sparse_categorical_accuracy: 0.8833 Epoch 67/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4304 - sparse_categorical_accuracy: 0.8417 Epoch 68/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4292 - sparse_categorical_accuracy: 0.8583 Epoch 69/100 4/4 [==============================] - 0s 750us/step - loss: 0.5901 - sparse_categorical_accuracy: 0.7083 Epoch 70/100 4/4 [==============================] - 0s 4ms/step - loss: 0.3781 - sparse_categorical_accuracy: 0.9500 Epoch 71/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3938 - sparse_categorical_accuracy: 0.9167 Epoch 72/100 4/4 [==============================] - 0s 1ms/step - loss: 0.3954 - sparse_categorical_accuracy: 0.9083 Epoch 73/100 4/4 [==============================] - 0s 3ms/step - loss: 0.4345 - sparse_categorical_accuracy: 0.8333 Epoch 74/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4539 - sparse_categorical_accuracy: 0.8250 Epoch 75/100 4/4 [==============================] - 0s 10ms/step - loss: 0.4821 - sparse_categorical_accuracy: 0.7917 Epoch 76/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3892 - sparse_categorical_accuracy: 0.9250 Epoch 77/100 4/4 [==============================] - 0s 15ms/step - loss: 0.3969 - sparse_categorical_accuracy: 0.8750 Epoch 78/100 4/4 [==============================] - 0s 3ms/step - loss: 0.5736 - sparse_categorical_accuracy: 0.7750 Epoch 79/100 4/4 [==============================] - 0s 3ms/step - loss: 0.3861 - sparse_categorical_accuracy: 0.9000 Epoch 80/100 4/4 [==============================] - 0s 47ms/step - loss: 0.3713 - sparse_categorical_accuracy: 0.9583 - val_loss: 0.3176 - val_sparse_categorical_accuracy: 1.0000 Epoch 81/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4649 - sparse_categorical_accuracy: 0.8167 Epoch 82/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4421 - sparse_categorical_accuracy: 0.8583 Epoch 83/100 4/4 [==============================] - 0s 2ms/step - loss: 0.5516 - sparse_categorical_accuracy: 0.7583 Epoch 84/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4063 - sparse_categorical_accuracy: 0.9083 Epoch 85/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3740 - sparse_categorical_accuracy: 0.9333 Epoch 86/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3908 - sparse_categorical_accuracy: 0.9417 Epoch 87/100 4/4 [==============================] - 0s 1ms/step - loss: 0.3956 - sparse_categorical_accuracy: 0.9417 Epoch 88/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3925 - sparse_categorical_accuracy: 0.9000 Epoch 89/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3834 - sparse_categorical_accuracy: 0.9167 Epoch 90/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4654 - sparse_categorical_accuracy: 0.7917 Epoch 91/100 4/4 [==============================] - 0s 2ms/step - loss: 0.4126 - sparse_categorical_accuracy: 0.9083 Epoch 92/100 4/4 [==============================] - 0s 1ms/step - loss: 0.6133 - sparse_categorical_accuracy: 0.7083 Epoch 93/100 4/4 [==============================] - 0s 1ms/step - loss: 0.4386 - sparse_categorical_accuracy: 0.8333 Epoch 94/100 4/4 [==============================] - 0s 2ms/step - loss: 0.3646 - sparse_categorical_accuracy: 0.9500 Epoch 95/100 4/4 [==============================] - 0s 3ms/step - loss: 0.3656 - sparse_categorical_accuracy: 0.9667 Epoch 96/100 4/4 [==============================] - 0s 1ms/step - loss: 0.3967 - sparse_categorical_accuracy: 0.9167 Epoch 97/100 4/4 [==============================] - 0s 2ms/step - loss: 0.5092 - sparse_categorical_accuracy: 0.7667 Epoch 98/100 4/4 [==============================] - 0s 3ms/step - loss: 0.3876 - sparse_categorical_accuracy: 0.9250 Epoch 99/100 4/4 [==============================] - 0s 4ms/step - loss: 0.4302 - sparse_categorical_accuracy: 0.8333 Epoch 100/100 4/4 [==============================] - 0s 90ms/step - loss: 0.3709 - sparse_categorical_accuracy: 0.8917 - val_loss: 0.3749 - val_sparse_categorical_accuracy: 0.9333 Model: "sequential" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense (Dense) multiple 15 ================================================================= Total params: 15 Trainable params: 15 Non-trainable params: 0 _________________________________________________________________
通过 model.summary 打印出我们搭建的神经网络的结构和参数统计,该网络结构为 4 个输入(4 个特征值),3 个输出(标签 3 分类)的一层网络(一个 w),dense 全连接共有 15 个参数(参数 w 是 4 行 3 列共 12 个元素,参数 b 是 3 个元素的向量,共 15 个参数),Trainable params 是可训练参数共有 15 个,Non-trainable 是不可训练参数有 0 个。
在类里面使用 keras 实现鸢尾花分类
下面我们在类里面使用 keras 的六步法编写代码来实现鸢尾花分类。
import tensorflow as tf from tensorflow.keras.layers import Dense from tensorflow.keras import Model from sklearn import datasets import numpy as np x_train = datasets.load_iris().data y_train = datasets.load_iris().target np.random.seed(1024) np.random.shuffle(x_train) np.random.seed(1024) np.random.shuffle(y_train) tf.random.set_seed(1024) class IrisModel(Model): def __init__(self): super(IrisModel, self).__init__() self.d1 = Dense(3, activation='softmax', kernel_regularizer=tf.keras.regularizers.l2()) def call(self, x): # 在call函数中调用self.d1实现了从输入x到出y的前向传播 y = self.d1(x) return y model = IrisModel() model.compile(optimizer=tf.keras.optimizers.SGD(lr=0.1), loss=tf.keras.losses.SparseCategoricalCrossentropy(from_logits=False), metrics=['sparse_categorical_accuracy']) model.fit(x_train, y_train, batch_size=32, epochs=500, validation_split=0.2, validation_freq=20) model.summary()
代码运行的结果和上面一致,通过 model.summary 打印出的网络结构也是一样的,大家可以自行试验。
如果神经网络有多层,我们只需要在构造函数中定义多个 Dense 即可,我们在 call 函数中调用相应的 Dense 实现前向传播,model.fit 中会不停的调用对象中的 call 函数(函数对象)。其余写法和我们前面一致。
sigmod 和 softmax 的区别
网上代码案例中同学们可能看到有的分类问题使用激活函数 sigmod 有的使用激活函数 softmax,总是分不清这两者有什么区别,在此我们就来讲解一下这两者的区别。
sigmoid=多标签分类问题=多个正确答案=非独占输出(例如胸部X光检查、住院)。构建分类器,解决有多个正确答案的问题时,用 sigmoid 函数分别处理各个原始输出值,sigmoid 函数是一种 logistic 函数,它将任意的值转换到 (0,1)之间,但这些值之间没有关联,都是属于独立分布。
softmax=多类别分类问题=只有一个正确答案=互斥输出(例如手写数字,鸢尾花)。构建分类器,解决只有唯一正确答案的问题时,用 softmax 函数处理各个原始输出值,它将任意的值转换到 (0,1)之间。softmax 函数的分母综合了原始输出值的所有因素,这意味着,softmax 函数得到的不同概率之间相互关联,而这些值的累和为1(满足概率的性质)。
如果模型输出为非互斥类别,且可以同时选择多个类别,则采用 sigmoid 函数计算该网络的原始输出值。如果模型输出为互斥类别,且只能选择一个类别,则采用 softmax 函数计算该网络的原始输出值。sigmoid 函数可以用来解决多标签问题,softmax 函数用来解决单标签问题。对于某个分类场景,当 softmax 函数能用时,sigmoid 函数一定可以用。
假设有一个前向传播的输出为:[-0.9419267177581787, 1.944047451019287],对应的类别分别为(0, 1),这个属于分类结果唯一,我们分别使用 sigmod 和 softmax 激活函数运算,代码如下:
import tensorflow as tf y = tf.constant([-0.9419267177581787, 1.944047451019287]) print(tf.nn.sigmoid(y)) print(tf.nn.softmax(y))
程序运行结果如下:
tf.Tensor([0.28051132 0.8747961 ], shape=(2,), dtype=float32) tf.Tensor([0.05285128 0.9471487 ], shape=(2,), dtype=float32)
我们经过 sigmoid 运算的结果为:[0.2805, 0.8748]。前者 0.2805 指的是分类类别为 0 的概率;0.8748 指的是分类类别为 1 的概率。二者相互独立,可看作两次独立的实验(显然在这里不适用,因为 0-1 类别之间显然不是相互独立的两次伯努利事件)。所以显而易见的,二者加和并不等于 1。
我们经过 softmax 运算的结果为 [0.0529, 0.9471],这里两者加和是 1,对于分类结果唯一的问题,我们使用 softmax 函数明显更精确。
假设有一个前向传播的输出为:[-0.9, 1.9, 0.3,1.5],对应的类别分别为(0, 1 , 0 , 1),这个属于分类结果不唯一,我们分别使用 sigmod 和 softmax 激活函数运算,代码如下:
import tensorflow as tf y = tf.constant([-0.9, 1.9, 0.3, 1.5]) print(tf.nn.sigmoid(y)) print(tf.nn.softmax(y))
程序运行的结果如下:
tf.Tensor([0.2890505 0.8698916 0.5744425 0.81757444], shape=(4,), dtype=float32) tf.Tensor([0.03145847 0.51732343 0.10444581 0.34677225], shape=(4,), dtype=float32)
从结果上看,使用 sigmod 的效果更好。
本节重要知识点
会使用 keras 搭建神经网络
会使用类的方法编写代码
了解 sigmod 和 softmax 使用场景





